Bioinformatics Toolbox
Bioinformatics Toolbox™ предоставляет алгоритмы и приложения для секвенирования следующего поколения (NGS, Next Generation Sequencing), анализа микрочипов ДНК, масс-спектрометрии и генной онтологии. Используя функции инструмента, осуществляется чтение геномных и протеомных данных из стандартных форматов файлов, таких как SAM, FASTA, CEL и CDF, а также из баз данных, таких как NCBI Gene Expression Omnibus и GenBank. Полученные данные могут быть исследованы с помощью браузеров последовательностей, пространственных тепловых карт и кластерных диаграмм. Инструмент также предоставляет статистические методы для обнаружения пиков, восстановления значений для отсутствующих данных и выбора признаков.
Функции инструмента могут быть скомбинированы для поддержки общих рабочих процессов биоинформатики. Данные ChIP-Seq могут быть использованы для определения факторов транскрипции, выполняется анализ данных RNA-Seq для идентификации дифференциально экспрессируемых генов, идентифицируются варианты количества копий и SNP в данных микроматрицы, с использованием данных масс-спектрометрии осуществляется классификация белковых профилей.
Ключевые особенности
-
Анализ последовательностей с помощью секвенирования следующего поколения и браузер результатов
-
Анализ и визуализация последовательностей, включая парное и множественное выравнивание последовательностей и обнаружение пиков
-
Анализ данных микрочипов, включая чтение, фильтрацию, нормализацию и визуализацию
-
Масс-спектрометрический анализ, включая предварительную обработку, классификацию и идентификацию маркеров
-
Филогенетический анализ дерева
-
Функции теории графов, включая карты взаимодействия, графики иерархии и пути
-
Импорт данных из геномных, протеомных и генных файлов экспрессии, включая SAM, FASTA, CEL и CDF, а также из баз данных, таких как NCBI и GenBank
Анализ секвенирования следующего поколения
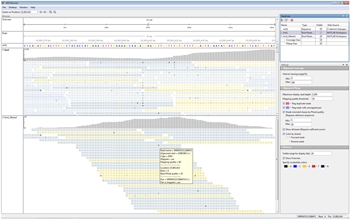
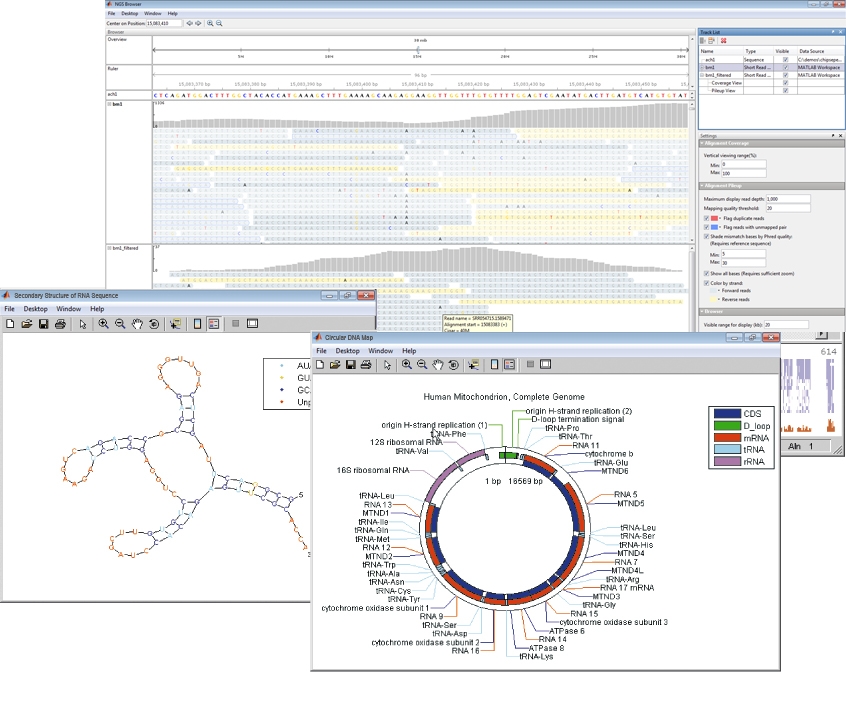
Bioinformatics Toolbox предоставляет алгоритмы и методы визуализации для анализа секвенирования следующего поколения. Bioinformatics Toolbox позволяет анализировать целые геномы при выполнении расчетов на уровне разрешения базовой пары. Браузер NGS используется для визуализации и исследования выравниваний краткого чтения с использованием односортного или парного короткого чтения. Также можно создавать собственные процедуры анализа, как показано в следующих примерах.
Визуализация и исследование выравнивания
Браузер NGS используется для проверок и исследования выравнивания коротко читаемых последовательностей для поддержки анализа, который измеряет генетические вариации и экспрессию генов. Браузер NGS позволяет:
-
Визуализацию кратко прочитанных данных, выровненных по нуклеотидной эталонной последовательности
-
Сравнение несколько наборов данных, выровненных по общей эталонной последовательности
-
Просмотр покрытия разных баз и областей эталонной последовательности
-
Изучение качества и других характеристик выровненных чтений
-
Выявление несоответствия из-за ошибок базового вызова или полиморфизмов
-
Визуализация вставок и удалений
-
Получение аннотаций объектов относительно определенной области эталонной последовательности
Хранение и управление кратко читаемыми данными последовательности
Наборы данных, используемые в анализе последовательности следующего поколения, часто слишком велики, чтобы поместиться в физическую память. Bioinformatics Toolbox предоставляет специализированные контейнеры данных, которые позволяют анализировать целые геномы.
Объект BioIndexedFile позволяет получить доступ к содержимому текстовых файлов, содержащих записи неодинакового размера, такие как последовательности, аннотации и перекрестные ссылки на набор данных. Эти объекты генерируются из таблиц, текстовых файлов или специальных форматов приложений, таких как SAM, FASTA и FASTQ.
Класс BioMap хранит информацию из кратко читаемых последовательностей, включая заголовки последовательностей, считываемые последовательности, показатели качества, а также данные о выравнивании и сопоставлении с одной эталонной последовательностью. Свойства и методы объекта используются для исследования, доступа, фильтрации и манипулирования данными, содержащимися в объекте BioMap.
Анализ и визуализация данных микрочипов
Bioinformatics Toolbox позволяет анализировать и понимать необработанные данные микрочипов.
Нормализация микрочипов
Доступно несколько методов для нормализации данных микрочипов, включая локально-линейную модель, глобальное среднее, медианное абсолютное отклонение (MAD) и квантильную нормализацию. Эти методы применимы как ко всему чипу микроматрицы, так и к определенным областям или блокам. Функции фильтрации и вменения позволяют очищать необработанные данные перед запуском процедур анализа и визуализации.
Анализ данных и визуализация
Bioinformatics Toolbox позволяет выполнять коррекцию фона и вычислять значения экспрессии гена (набора зондов) из данных уровня зондов микрочипов Affymetrix , используя процедуры Robust Multi-Array Average (RMA) и GC Robust Multi-Array Average (GCRMA). Можно применить круговую двоичную сегментацию к массиву данных CGH и оценить частоту ложных обнаружений при проверке множественных гипотез данных экспрессии генов из эксперимента с микрочипами. Также можно выполнить рангово-инвариантную нормализацию набора либо для интенсивностей зондов для нескольких файлов Affymetrix CEL, либо для значений экспрессии генов из двух разных условий эксперимента.
Специализированные процедуры для визуализации данных микрочипов включают в себя графики вулканов, графики «ящик с усами», диаграммы журналов , ИК-диаграммы и пространственные тепловые карты микрочипа. Также можете визуализировать идеограммы с помощью шаблонов G-полос.
С помощью Statistics and Machine Learning Toolbox™ можно выполнить классификацию результатов, выполнить иерархическую кластеризацию и кластеризацию по методу K-средних значений и представить данные микрочипов в статистических визуализациях, таких как 2D-кластерные диаграммы с оптимальным упорядочением листьев, тепловые карты, графики основных компонентов и деревья классификации.
Анализ данных масс-спектрометрии
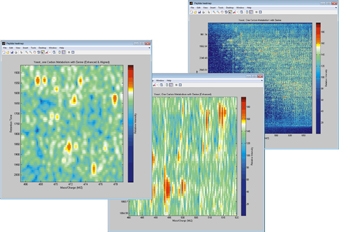
Bioinformatics Toolbox предоставляет набор функций для анализа данных масс-спектрометрии. Эти функции позволяют выполнять предварительную обработку, классификацию и идентификацию маркеров по данным SELDI, MALDI, LC/MS и GC/MS. Функции предварительной обработки включают базовую коррекцию, сглаживание, калибровку и повторную выборку. Необработанные данные спектров выравниваются, используя ось M/Z, и выполняется выравнивание по времени удержания для данных LC/MS и GC/MS. Доступно одновременное построение нескольких спектров.
После сглаживания, выравнивания и нормализации спектров можно использовать инструменты классификации и машинного обучения для создания классификаторов и выявления потенциальных биомаркеров.
Дифференциальный протеомный и метаболомический анализ без меток с использованием Bioinformatics Toolbox.
Теория графов, статистическое обучение и генная онтология
Теория графов и визуализация
Bioinformatics Toolbox позволяет применять базовую теорию графов для разреженных матриц. Доступно создание, просмотр и управление графами, такими как карты взаимодействия, графики иерархии и пути. Доступны определение и просмотр кратчайших путей в графах, проверка на циклы в направленных графах и нахождение изоморфизма между двумя графами.
Машинное обучение и визуализация
Bioinformatics Toolbox предоставляет функции, основанные на алгоритмах классификации и статистического обучения в Statistics and Machine Learning Toolbox, в том числе:
-
Классификатор опорных векторов (SVM) и K-ближайший сосед
-
Функции для постановки экспериментов по перекрестной проверке и измерения эффективности различных методов классификации
-
Интерактивные инструменты для выбора объектов, отображения и отображения графиков и путей иерархии
Генная онтология
Bioinformatics Toolbox позволяет получить доступ к базе данных онтологии генов из MATLAB, проанализировать аннотированные файлы генной онтологии и получить подмножества онтологии, такие как предки, потомки или родственники.
Анализ последовательностей
Bioinformatics Toolbox предоставляет инструменты анализа и визуализации последовательностей для данных геномных и протеомных последовательностей. Доступны различные виды анализа, включая множественные выравнивания последовательностей, а также создание и интерактивный просмотр и манипулирование филогенетическими деревьями.
Выравнивание последовательности
Bioinformatics Toolbox предоставляет функции, объекты и методы для анализа последовательностей, включая попарную последовательность, профиль последовательности и выравнивание нескольких последовательностей. Они включают:
-
Реализацию стандартных алгоритмов MATLAB для локального и глобального выравнивания последовательностей, таких как алгоритмы модели Нидлмана-Вунша, Смита-Уотермана и Маркова, скрытые по профилю
-
Прогрессивное выравнивание нескольких последовательностей
-
Графическое представление матриц результатов выравнивания
-
Стандартные оценочные матрицы, такие как семейства матриц PAM и BLOSUM
-
Расчет консенсусной последовательности и отображение логотипа последовательности
Утилиты и статистика
Bioinformatics Toolbox позволяет манипулировать и анализировать последовательности, чтобы глубже понять данные. Доступно:
-
Преобразование последовательностей ДНК или РНК в аминокислотные последовательности с использованием генетического кода
-
Выполнение статистического анализа последовательностей и нахождение конкретных шаблонов в последовательностях
-
Применение эндонуклеазов рестрикции и протеазы для выполнения расщепления последовательностей in-silico или создания случайных последовательности для тестов
-
Предсказание минимальной свободной энергии вторичной структуры последовательностей РНК
Визуализация последовательностей
Bioinformatics Toolbox позволяет визуализировать последовательности и выравнивания. Доступны линейные или круговые карты последовательностей, аннотированных функциями GenBank. Доступна визуализация вторичных структурных схем последовательностей РНК. Интерактивные средства просмотра позволяют исследовать и изменять парные и множественные выравнивания последовательностей.
Филогенетический анализ дерева
Bioinformatics Toolbox позволяет создавать и редактировать филогенетические деревья. Доступен расчет попарных расстояний между выровненными или невыровненными нуклеотидными или аминокислотными последовательностями с использованием широкого диапазона метрик сходства, таких как модель Джукса-Кантора, p-расстояние, оценка выравнивания или метод определения расстояния, определенный пользователем. Филогенетические деревья строятся с использованием иерархической связи с различными методами, включая соединение соседей, одиночную и полную связь, а также среднее значение метода групп невзвешенных пар (UPGMA).
Инструмент поддерживает взвешивание и смену корня деревьев, вычисление поддеревьев и вычисление канонической формы деревьев. Средство просмотра филогенетического дерева позволяет обрезать, переупорядочивать и переименовывать ветви, исследовать расстояния; обеспечивает чтение или запись файлов в формате Newick. Возможности построения графики MATLAB обеспечивают создание деревьев высокого качества.
Анализ свойств белка
Bioinformatics Toolbox предоставляет методы анализа последовательности белка, включая процедуры для расчета свойств последовательности пептида, таких как атомный состав, изоэлектрическая точка и молекулярный вес. Инструмент предоставляет возможности определения аминокислотного состава белковых последовательностей, расщепления белков с помощью ферментов и создания карт Рамачандрана для данных PDB. Sequence Tool используется для просмотра свойств аминокислотной последовательности, Molecule Viewer используется для отображения и управления трехмерными молекулярными структурами.
Импорт данных и развертывание приложений
Форматы файлов и доступ к базам данных
Осуществляется доступ к стандартным форматам файлов для биологических данных, онлайн-баз данных и веб-сайтов. Bioinformatics Toolbox позволяет:
-
Чтение данных последовательности из стандартных форматов файлов, включая FASTA, PDB и SCF
-
Чтение данных микрочипов из файловых форматов, таких как файлы Affymetrix DAT, EXP, CEL, CHP и CDF; формат данных результатов ImaGene; файлы программного обеспечения Agilent Feature Extraction; файлы GenePix GPR и GAL
-
Чтение данных из онлайн-баз данных, таких как GenBank, EMBL, NCBI BLAST и PDB
-
Импорт данных непосредственно с веб-сайта NCBI Gene Expression Omnibus с помощью одной команды
-
Чтение информации о цитогенетических полосах из идеограмм NCBI или текстовых файлов цитобанда UCSC
-
Получение данных масс-спектрометрии из файлов MZXML и JCAMP-DX
Совместное использование алгоритмов и развертывание приложений
MATLAB предоставляет инструменты, которые позволяют превратить пользовательскую программу анализа данных в специализированное программное приложение. К ним относятся инструменты разработки для создания пользовательских интерфейсов, визуальная интегрированная среда разработки и профилировщик. Продукты MATLAB для развертывания приложений позволяют интегрировать алгоритмы MATLAB с существующим C, C++ и Java ™ кодом приложений и обеспечивают развертывание разработанных алгоритмов и пользовательских интерфейсов в виде автономных приложений, преобразующие алгоритмы MATLAB в сборки Microsoft .NET или COM-компонентов , которые могут быть доступны из любого COM-приложения и создавать надстройки Microsoft Excel.
Также поддерживается интегрирация MATLAB с широко используемыми инструментами биоинформатики, такими как BioPerl, веб-сервисами на основе SOAP и COM-плагинами.
Передача аргументов из MATLAB в скрипты Perl и перенос данные поиска BLAST обратно в MATLAB.
 Документация
Документация Изучение узлов связывания белок-ДНК по данным парного конца ChIP-Seq
Изучение узлов связывания белок-ДНК по данным парного конца ChIP-Seq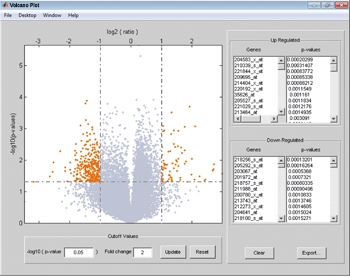
 Statistics and Machine Learning Toolbox
Statistics and Machine Learning Toolbox
